Oligonucleotidi antisenso allele specifici come possibile approccio terapeutico nell’Atassia Spinocerebellare di tipo 1 (SCA1)
Alfredo Prof. Brusco
Oligonucleotidi antisenso allele specifici come possibile approccio terapeutico nell’Atassia Spinocerebellare di tipo 1 (SCA1)
Le atassie spinocerebellari (SCA) sono un gruppo di malattie autosomiche dominanti eterogenee dal punto di vista clinico e genetico, caratterizzate da andatura instabile, difficoltà di coordinazione dei movimenti degli arti e problemi nell’articolazione della parola. Questi segni di natura cerebellare possono essere associati ad altri segni neurologici, come oftalmoplegia e/o declino cognitivo. La prevalenza delle SCA è stimata in 1-3/100.000 soggetti, con significative variazioni di tipo geografico ed etnico.
Una delle Atassie Spinocerebellari più frequenti in Italia è la SCA1, la cui causa è determinata dall’ espansione di una ripetizione CAG nella regione codificante dell’esone 8 del gene ATXN1. L’aumento nel numero delle triplette CAG, che codifica normalmente per l’aminoacido glutammina (Q), impedisce il corretto funzionamento della proteina ataxin-1. I dati di letteratura suggeriscono che nel cervelletto umano le proteine mutate, ricche di tratti polyQ, formano aggregati che accumulandosi danneggiano le cellule del Purkinje.
La maggior parte delle strategie terapeutiche ad oggi proposte per le patologie da polyQ sono mirate a correggere uno dei processi cellulari alterati dall’espressione della proteina mutata.
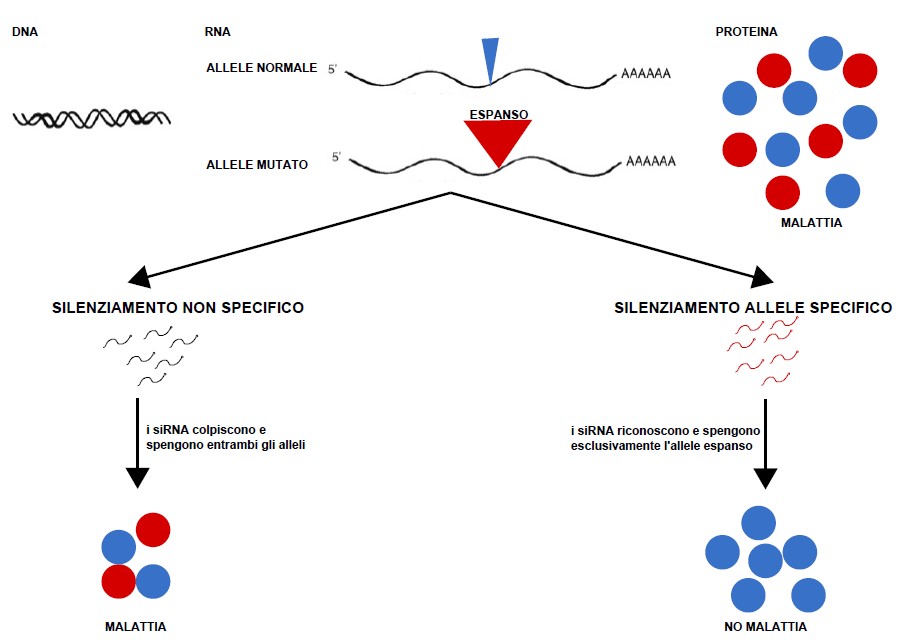
La riduzione dei livelli di proteina Atxn1-polyQ e quindi l’annullamento degli effetti tossici a livello cellulare, potrebbero rappresentare un metodo efficace rispetto a quello mirato a correggere una singola via di segnale. Un possibile approccio per raggiungere questo obiettivo, consiste nello spegnere il gene di interesse tramite l’RNA interference (RNAi), un processo fisiologico utilizzato normalmente dalla cellula per modulare l’espressione genica o come meccanismo di difesa da agenti patogeni. L’espressione del gene può essere modulata attraverso l’introduzione nella cellula di piccole molecole sintetiche RNA doppio filamento (Small interfering RNA –siRNA) in grado di riconoscere e legare una specifica sequenza di RNA messaggero causandone la degradazione. Recentemente è stato dimostrato che l’RNAi può essere allele specifico, ovvero mutazione specifico.
Il silenziamento allele-specifico tramite RNAi (ASP-RNAi) consiste nell’inibire in modo specifico l'espressione dell’allele contenente l’espansione che causa la malattia, riducendo al minimo gli effetti sull’allele non espanso (Fig. 1). Per questo approccio è necessario identificare una variazione nucleotidica, definita polimorfismo a singolo nucleotide (SNP), che sia presente sull’allele mutato, ovvero in linkage con l’espansione CAG. L’efficacia di questo metodo è stata dimostrata in una linea di fibroblasti di un paziente SCA1 provando la sua possibile applicazione terapeutica in questa patologia (Fiszer et al. 2012). Questo tipo di metodica permette di conservare intatta la sintesi della proteina sana, fondamentale nel mantenimento delle normali funzioni cellulari.
Il progetto ha come obiettivo il verificare la fattibilità e l’efficacia del silenziamento allele-specifico come possibile terapia per la SCA1 sfruttando un modello cellulare reporter generato dal nostro laboratorio e successivamente in linee di fibroblasti SCA1.
Figura 1. Razionale dell’approccio di silenziamento “allele specifico”.
Gruppo di ricerca
