Studio della patogenesi dell’Atassia Spinocerebellare tipo 38 (SCA38)
Alfredo Prof. Brusco
Studio della patogenesi dell’Atassia Spinocerebellare tipo 38 (SCA38)
L’atassia spinocerebellare di tipo 38 (SCA38) è una rara forma di atassia, la cui incidenza è stata stimata intorno al 1% fra le forme autosomiche dominanti di atassia. Questa patologia ha un’insorgenza nell’adulto intorno alla quarta decade di età, ed è caratterizzata da un fenotipo cerebellare puro con una progressione clinica lenta. All’esordio la patologia è contraddistinta da nistagmo ed atassia della marcia, che si associa frequentemente a pes cavus ed iposmia. All’analisi della risonanza magnetica i pazienti mostrano un’atrofia del cervelletto senza un coinvolgimento della sostanza bianca.
Abbiamo identificato come gene causative di questa forma di SCA il gene ELOVL5 che codifica per un enzima coinvolto nella sintesi degli acidi grassi a lunga catena appartenenti alla famiglia degli omega-3 e omega -6, in particolare dell’acido docoasesanoico (DHA, Ω3), l’acido eicosapentaenoico (EPA, Ω3) e l’acido arachidonico (Ω6). Questo enzima è espresso ubiquitariamente e nel cervelletto mostra un’espressione specifica nelle cellule del Purkinje, tessuto che va incontro a degenerazione nelle SCA.
Le mutazioni attualmente identificate sono due sostituzioni amminoacidiche missense (p.Leu72Val e p.Gly230Val), di cui la p.Leu72Val è presente in una singola famiglia francese e la p.Gly230Val che è condivisa da tre famiglie Italiane ( (Di Gregorio et al. 2014).
La mutazione p.Gly230Val altera la funzione dell’enzima causando una riduzione dei suoi prodotti di sintesi nel siero dei pazienti SCA38 analizzati (Fig. 1a). L’espressione di questo gene è finemente regolata dalla quantità dei suoi prodotti di sintesi attraverso un sistema di regolazione a feedback; ELOVL5, infatti, ha un aumento di espressione sia a livello di RNA messaggero (Fig. 1b) sia a livello proteico (Fig. 1c) nei linfoblasti dei pazienti in cui è stato dimostrata una riduzione degli acidi grassi da lui sintetizzati.
Abbiamo inoltre dimostrato in diversi modelli cellulari esprimenti ELOVL5 con un tag-HALO che entrambe le mutazioni identificate alterano la normale localizzazione subcellulare della proteina, che non mostra, come atteso, un segnale diffuso nell’reticolo endoplasmatico, ma si concentra nella regione perinucleare e nell’apparato del Golgi della cellula (Fig 2 a e b).
Fig 2. Distribuzione della proteina ELOVL5 in cellule COS7 trasfettate con un vettore plasmidico esprimente o la proteina wild type (controllo) o quella mutata (p.Leu72Val e p. Gly230Val). Nel pannello a) e b) il segnale della proteina ELOVL5 è in rosso, mentre quello relativo alla calreticulina (proteina specifica del reticolo endoplasmatico) o di GM130 (proteina specifica dell’apparato del Golgi) è in verde.
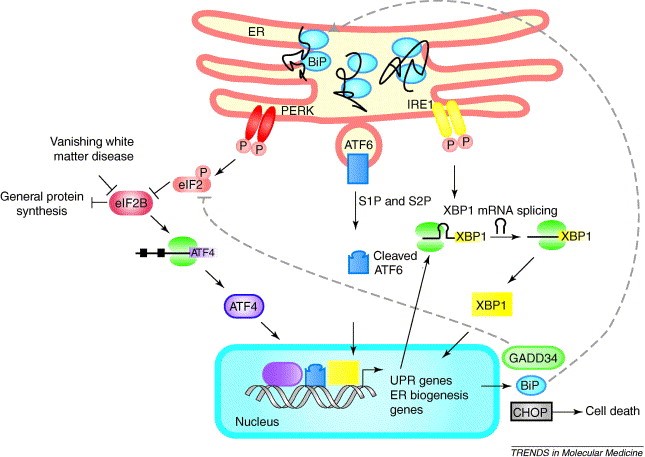
L’ alterata localizzazione della proteina mutata nei modelli cellulari analizzati, ci ha fatto ipotizzare che le mutazioni identificate potessero alterare la normale conformazione della proteina e che il suo accumulo nella regione perinucleare del reticolo endoplasmatico e nell’apparato del Golgi potesse indurre uno stress del reticolo endoplasmatico con conseguente attivazione della via nota come unfolded protein response (UPR)(Fig. 3).
Nel nostro studio ci proponiamo di testare questa ipotesi valutando l’espressione a livello proteico di alcuni geni che codificano per proteine coinvolte nell’UPR, quali ATF4, XBP1 e CHOP.
Inoltre ci proponiamo di valutare l’impatto delle mutazioni sulla conformazione della proteina trattando le cellule disponibili dei pazienti SCA38 con l’ inibitore del proteosoma MG-132 e con lo chaperone chimico fenilbutirrato, noto per stabilizzare le proteine con un’alterata conformazione. Qualora le sostituzioni amminoacidiche identificate alterassero la struttura conformazionale della proteina noi attendiamo un aumento di ELOVL5 a livello proteico nelle cellule dei pazienti trattate con MG-132 ed una sua riduzione in quelle trattate con il fenilbutirrato. Questi esperimenti potrebbero fornire delle informazioni sul meccanismo patogenetico chiarendo se le mutazioni identificate causino un accumulo della proteina alterata conferendogli una funzione tossica.
^Di Gregorio E, Borroni B et al: ELOVL5 Mutations Cause Spinocerebellar Ataxia 38. Am J Hum Genet 2014, 95(2):209-217.
Gruppo di Ricerca
